Zakończone projekty badawcze
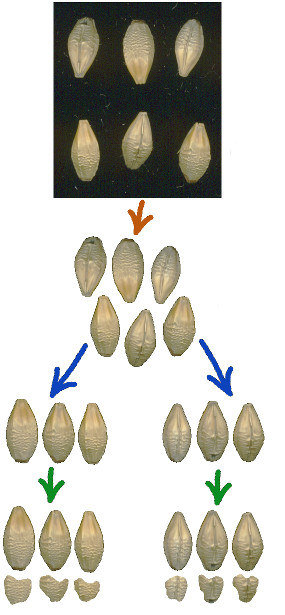
2015-2018: NCBR PBS3/A8/38/2015 Opracowanie przemysłowej metody automatycznej oceny parametrów technologicznych i klasyfikacji ziarna z zastosowaniem analizy obrazów. Projekt wykonywany był w konsorcjum: Uniwersytet Warmińsko-Mazurski w Olsztynie (lider), Politechnika Łódzka, Politechnika Wrocławska i Słodownia Soufflet Polska Sp. z o.o. Celem projektu było opracowanie automatycznej kompleksowej technologii oceny jakości mieszaniny ziarna dla przemysłu browarniczego.
2015-2017: Grant NCN ST7/OPUS-8 Rozwój numerycznych metod modelowania oraz oceny perfuzji nerek z użyciem obrazowania rezonansu magnetycznego. Kierownikiem projektu był dr Artur Klepaczko. Celem było udoskonalenie metod pomiaru parametrów perfuzji na podstawie obrazów angiografii rezonansu magnetycznego oraz opracowanie symulatora obrazowania perfuzji nerek.
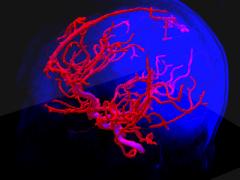
2013-2016: NCN 2013/08/M/ST7/00943 Numeryczne modelowanie układu żył i tętnic mózgu w skali makro- i mezoskopowej na podstawie trójwymiarowych obrazów rezonansu magnetycznego – projekt kierowany przez prof. dr hab. inż. Andrzeja Materkę. Udział w eksperymentach segmentacji obrazów rezonansu magnetycznego układu naczyń krwionośnych. Opracowanie i integracja algorytmów segmentacji oraz modelowania układu krwionośnego na podstawie angiogramów.
2011-2014: N313 789140 - Wykorzystanie ultradźwięków oraz komputerowej analizy obrazu do oceny jakości mięsa i wędlin. Projekt był kierowany przez dr. Piotra Zapotocznego z Uniwersytetu Warmińsko Mazurskiego. W ramach projektu zająłem się segmentacją obrazów wędlin i obliczaniem ich cech na podstawie obrazów barwnych. Cechy obrazowe były porównywane z wartościami pomiarów fizyko-chemicznych. Celem projektu było opracowanie taniej i szybkiej metody do oceny jakości wędlin.
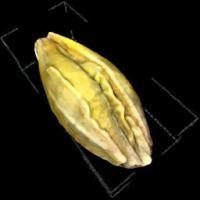
2010-2013: 4498/B/P01/2010/39 - Opracowanie inteligentnego systemu wizyjnego do identyfikacji odmian oraz właściwości fizycznych ziarna jęczmienia browarnego. W ramach współpracy z Uniwersytetem Warmińsko Mazurskim opracowałem metodę analizy obrazów ziarniaków zbóż, która umożliwia identyfikację, określenie orientacji i wyznaczenie obszaru pofałdowanego pojedynczych ziaren. Powstała również metoda do wizyjnego oznaczania przynależność odmianowej ziarniaków.
W latach 2011-2013 brałem udział w projekcie 6509/B/T02/2011/40 Wspomagany sprzętowo symulator angiografii rezonansu magnetycznego do walidacji metod ilościowej analizy układu naczyń krwionośnych finansowanym przez Ministerstwo Nauki i Szkolnictwa Wyższego, i kierowanym przez dr. Artura Klepaczko. Moim udziałem było opracowanie metody śledzenia ruchu małych objętości płynu na potrzeby symulacji zjawisk rezonansu magnetycznego w obrazowaniu angiograficznym. Metoda umożliwiła rozdzielenie symulacji przepływu płynu od symulacji zjawisk rezonansu magnetycznego. Dotychczas oba elementy kompletnej symulacji musiały być prowadzone równolegle w tym samym czasie.
W latach 2010 wziąłem udział w projekcie R02 01303 finansowanym przez MNiSW i kierowanym przez prof. Pawła Strumiłło. Jednym z przedsięwzięć projektu było opracowanie systemu wspomagania osób niepełnosprawnych z wykorzystaniem stereowizji i koncepcji tzw. udźwiękowienia sceny. Przygotowałem koncepcję integracji takiego systemu z zastosowaniem technologii DirecShow oraz wziąłem udział w integracji procedur opracowanych przez poszczególnych wykonawców.
W latach 2008–2010 kierowałem projektem badawczym N N518 326335 Wspomaganie diagnostyki medycznej przewodu pokarmowego z zastosowaniem analizy komputerowej sekwencji obrazów uzyskiwanych z endoskopu kapsułkowego finansowanego z funduszu MNiSW. Celem projektu było zbadanie przydatności cech teksturowych i barwowych do detekcji wybranych zmian patologicznych w obrazach z endoskopu kapsułkowego. Prace prowadziłem wraz z zespołem specjalistów z Instytutu Elektroniki Politechniki Łódzkiej, we współpracy z lekarzami Uniwersytetu Medycznego w Łodzi, dr. Piotrem Danielem i dr. Markiem Pazurkiem.
W latach 2007–2010 brałem udział w projekcie międzynarodowym DFG/36/2007 Opracowanie szybkich nieinwazyjnych sekwencji angiografii rezonansu magnetycznego do segmentacji oraz ilościowej analizy układu naczyń tętniczych i żylnych mózgu na obrazach 3D dużej rozdzielczości. Do moich zadań należało opracowanie metod segmentacji obrazów angiograficznych.
W latach 2004–2007 brałem udział w projekcie SPUB 138/E-370/SPB/COST/T-11/DWM60 Modelowanie obrazów tomograficznych rezonansu magnetycznego z uwzględnieniem procesów fizjologicznych i struktury fizycznej obrazowanych tkanek.
W latach 2002–2004 jako stypendysta Ministerstwa Handlu USA (Department of Commerce) brałem udział w pracach badawczych prowadzonych w National Institute of Standards and Technology (NIST) w Gaithersburgu. Zajmowałem się tam zagadnieniami projektowania systemów eksperckich. Moim zadaniem było opracowanie zintegrowanego środowiska programistycznego do tworzenia obiektowo zorientowanych systemów eksperckich o architekturze rozproszonej. Prace prowadziłem pod kierunkiem dr. Rama D. Srirama.
Podczas mojego pobytu w NIST miałem okazję poznać dr. D. Nageshwara Reddyego z Asian Institute of Gastroenterology (AIG). Dzięki niemu zainteresowałem się problematyką analizy filmów z endoskopu kapsułkowego. We współpracy z dr. Parupudim V.J. Sriramem z AIG rozpocząłem prace nad metodą rekonstrukcji obrazu wewnętrznej powierzchni przewodu pokarmowego na podstawie danych wideo.

W latach 1998–2001 brałem udział w projekcie europejskim COST-B11 Quantitation of Magnetic Resonance Image Texture, a następnie w latach 2003–2007 w projekcie COST B21 Physiological Modelling of MR Image Formation. Jednym z moich zadań było opracowanie systemu komputerowego do analizy tekstury w obrazach tomograficznych rezonansu magnetycznego. W wyniku tych prac powstał program o nazwie MaZda. Program MaZda został zaprezentowany na konferencjach, w publikacji książkowej Texture Analysis for Magnetic Resonance Imaging będącej podsumowaniem projektu COST B11 i B21, a przeglądowy artykuł dotyczący zastosowań programu i zaimplementowanych w nim metod został opublikowany w czasopiśmie Computer Methods and Programs in Biomedicine.
 W ramach grantu promotorskiego 8T11C02017 Identyfikacja obiektów w obrazach cyfrowych z zastosowaniem elastycznego modelu prowadziłem badania dotyczące rozwoju i zastosowań modeli deformowalnych w analizie obrazu. Modele siatek o różnych konfiguracjach połączeń międzywęzłowych zastosowałem do rozpoznawania i śledzenia obiektów będących w ruchu. Prowadziłem również prace dotyczące zastosowania modeli deformowalnych powierzchni do segmentacji obiektów trójwymiarowych w trójwymiarowych obrazach ultrasonograficznych i tomografii rezonansu magnetycznego. Wynikiem tych prac jest rozprawa doktorska prowadzona pod kierunkiem prof. Andrzeja Materki.
W ramach grantu promotorskiego 8T11C02017 Identyfikacja obiektów w obrazach cyfrowych z zastosowaniem elastycznego modelu prowadziłem badania dotyczące rozwoju i zastosowań modeli deformowalnych w analizie obrazu. Modele siatek o różnych konfiguracjach połączeń międzywęzłowych zastosowałem do rozpoznawania i śledzenia obiektów będących w ruchu. Prowadziłem również prace dotyczące zastosowania modeli deformowalnych powierzchni do segmentacji obiektów trójwymiarowych w trójwymiarowych obrazach ultrasonograficznych i tomografii rezonansu magnetycznego. Wynikiem tych prac jest rozprawa doktorska prowadzona pod kierunkiem prof. Andrzeja Materki.
W latach 1996–1997 brałem udział w projekcie badawczym 4P05D06812 Wspomagania diagnostyki raka sutka na podstawie komputerowej analizy mammogramów kierowanym przez dr. Janusza Racza. Zaimplementowałem procedury do obliczania cech teksturowych obrazów oraz dokonałem integracji procedur obliczeniowych przygotowanych przez pozostałych uczestników projektu. W wyniku tych prac powstał program o nazwie Mammo służący do klasyfikacji obrazów mammograficznych pod kątem uwidocznionych w nich zmian patologicznych.
W latach 1996–1998 w ramach projektu TEMPUS SJEP 09883-95 Education in Modern Microwave Techniques for Telecommunication - Restructuring and Development of New Courses w Institute für Hochfrequenztechnik, Technische Universität Berlin zająłem się problematyką komputerowej symulacji działania układów mikrofalowych z liniami transmisyjnymi koplanarnymi i szczelinowymi oraz sprzęgaczami tych linii. Nabyte doświadczenia i wiedzę wykorzystałem podczas organizowania laboratorium dydaktycznego Układy Częstotliwości Radiowych uruchomionego w Instytucie Elektroniki na przełomie 1997 i 1998 roku.
W latach 1996–1998 zajmowałem się analizą obrazów rentgenowskich ziaren zbóż. Prace były prowadzone w ramach projektu 5P06F01209 Fizyczne i biologiczne skutki suszenia mikrofalowego ziaren pszenicy we współpracy z dr. Jerzym Niewczasem z Instytutu Agrofizyki PAN w Lublinie. Wynikiem było opracowanie modeli deformowalnych do segmentacji, rozpoznawania orientacji, rzutu oraz określania położenia bruzdki i zarodka w ziarnach pszenicy.